MicroRNA (miRNA) 是一类长度约22nt的小非编码RNA,它们通过碱基互补配对的形式与mRNA结合,并降解或抑制靶mRNA的翻译。相比仅有几小时生命的mRNA,miRNA则更稳定,能维持超过一天甚至有时超过一周的稳定【1-3】。通常认为miRNA超凡的稳定性是由它们结合的AGO蛋白维持的。AGO蛋白可保护miRNA免受核酸外切酶的水解【4】。
有趣的是,有一部分miRNA的半衰期很短,小于2-10小时。这部分miRNA与靶mRNA有非常高的互补匹配度,而不限于5’端的种子序列(seed sequence)。因此,这一现象被称为靶标指导的miRNA降解(target-directed miRNA degradation, TDMD)。然而,目前对TDMD的具体机制尚不清楚。
近日,来自德州大学西南医学中心的Joshua T. Mendell教授和霍华德·休斯医学研究所的David P. Bartel教授分别在Science发表研究 A ubiquitin ligase mediates target-directed microRNA decay independently of tailing and trimming 和The ZSWIM8 ubiquitin ligase mediates target-directed microRNA degradation,这两项研究同时揭示了ZSWIM8通过泛素-蛋白酶体途径降解miRNA的机制。


CYRANO是一个非常保守的长链非编码RNA (lncRNA) ,只包含一个miR-7的结合位点,但具有非常高的匹配度(图1)。miR-7结合CYRANO后会很快被降解,属于TDMD范畴。
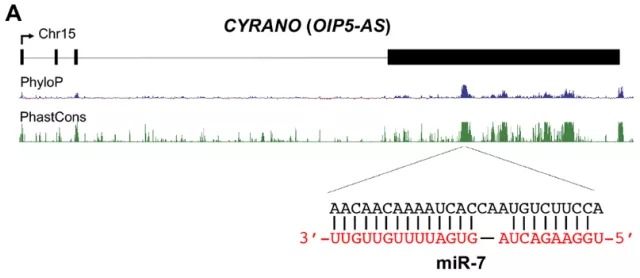
图1
为研究TDMD的具体机制,研究者利用CYRANO设计了报告基因,通过CRISPR screening的方法筛选参与TDMD的基因。两项研究同时发现敲低ZSWIM8可以解除CYRANO触发的miR-7降解,即显著地提高miR-7的表达。那么,ZSWIM8是否广泛地参与TDMD过程呢?研究者发现敲低ZSWIM8能够显著提高一批miRNA的表达,说明ZSWIM8能够介导TDMD过程;同时,该结果也鉴定了新的受TDMD调控的miRNA。
ZSWIM8是cullin-RING泛素连接酶(CRL)复合体的结合蛋白,参与蛋白的泛素-蛋白酶体降解途径。研究者发现ZSWIM8可以结合AGO2,促进AGO2的降解。
前人研究表明,当miRNA与靶基因高度匹配时,AGO2的蛋白构象会发生改变,暴露miRNA的3’末端。在这两项研究中,研究者发现ZSWIM8可以结合构象改变的AGO2蛋白,这也是为什么只有当miRNA与特定靶基因匹配时,才会触发TDMD现象(图2)。
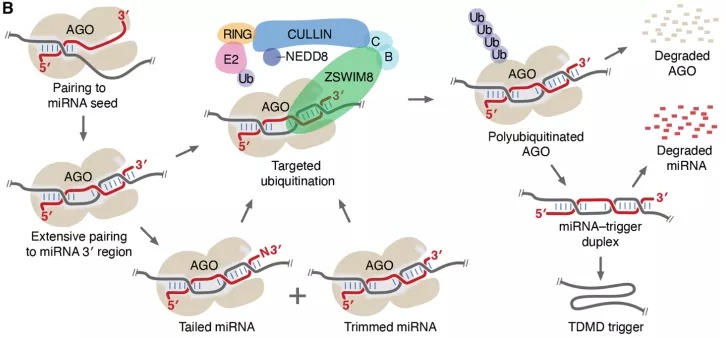
有趣的是,尽管TDMD现象已被发现了十多年,但以往的研究主要集中在miRNA的加尾和修剪(target-directed tailing and trimming, TDTT)中,而这两项研究从蛋白降解的角度揭示了miRNA降解的新机制。
总的来说,这两项研究通过高通量筛选的方法发现了ZSWIM8通过泛素-蛋白酶体途径降解AGO2蛋白和miRNA的机制,从新的角度揭示了TDMD现象的发生,具有重要意义。值得注意的是,ZSWIM8在线虫和果蝇中也有同源蛋白,提示ZSWIM8介导的TDMD在进化可能非常保守。同时,虽然TDMD被证明可以显著调控特定miRNA的表达,但是TDMD在发育和生理状态中的功能和意义一直未被阐明,这两项研究鉴定的ZSWIM8将提供特异性调控TDMD过程的工具。
参考文献
2. B. Reichholf, V. A. Herzog, N. Fasching, R. A. Manzenreither, I. Sowemimo, S. L. Ameres, Time-Resolved Small RNA Sequencing Unravels the Molecular Principles of MicroRNA Homeostasis. Mol. Cell 75, 756–768.e7 (2019). doi:10.1016/j.molcel.2019.06.018pmid:31350118
3. T. J. Eisen, S. W. Eichhorn, A. O. Subtelny, K. S. Lin, S. E. McGeary, S. Gupta, D. P. Bartel, The Dynamics of Cytoplasmic mRNA Metabolism. Mol. Cell 77, 786–799.e10 (2020). doi:10.1016/j.molcel.2019.12.005pmid:31902669
4. J. Sheu-Gruttadauria, P. Pawlica, S. M. Klum, S. Wang, T. A. Yario, N. T. Schirle Oakdale, J. A. Steitz, I. J. MacRae, Structural Basis for Target-Directed MicroRNA Degradation. Mol. Cell 75, 1243–1255.e7 (2019). doi:10.1016/j.molcel.2019.06.019pmid:31353209













